Quasi-SVM
SVM是一个强有力的机器学习算法,很多算法(比如RCNN)的分类器也使用SVM作为分类器,帮助我获得了博弈杯准确率排行榜的第二名(我使用的是VGG+Quasi-SVM。
但困难的是,如何把SVM和神经网络合并起来,且共同训练。于是有了近似SVM,我们可以使用hingeloss。
更幸运的是,keras提供了另一种方法,Random Fourier Features。
链接:
https://keras.io/examples/keras_recipes/quasi_svm
2025年更新: keras现在已经删掉了RandomFourierFeatures
Random Fourier Features
SVM的核心是核技巧和最大间隔,SVM具体实现我们不再赘述。
Random Fourier Features出自2007 年的《Random Features for Large-Scale Kernel Machines》,并于十年后获得了NeurIPS 2017的Test of Time Award。
他们的想法是使用一个随机化的特征图 $z: \mathbb{R}^D \to \mathbb{R}^d$ 来近似核函数 $k(x,y)$:
$$
k(x,y) = \langle \phi(x), \phi(y) \rangle_V \approx z(x)^\top z(y)
$$
这里,$V$ 是一个(可能是无限维的)希尔伯特空间,$D$ 是输入空间的维度,$d$ 是随机特征空间的维度。关键在于,如果 $d \ll N$,那么在特征空间中工作的计算成本要低得多。
根据 作者的blog,它的想法受到了以下观察的启发,设w是一个随机的D维向量,$w\sim N_D(0,I)$
定义$h:x\to exp(iw^Tx)$
则
$$
\begin{align}
E_w[h(x)h(y)^*]&=E_w[exp(iw^T(x-y))]
\\
&=\int_{R^D}p(w)exp(iw^T(x-y))dw
\\
&=exp(-\frac{1}{2}(x-y)^T(x-y))
\end{align}
$$
即其期望值为高斯核。
实际上它是一个Bochner 定理(Rudin,1962 年)——更一般结果的特定实例,即
Bochner 定理:一个在 $R^D$ 上的连续核$k(x,y)=k(x-y)$是正定的,当且仅当$k(\Delta)$ 是一个非负测度的傅里叶变换。
非负测度的傅里叶变换是$\int p(w)exp(iw\Delta)dw$
故有
$$
\begin{align}
k(x,y) &= k(x-y)
\\ &= \int p(w) \exp(i w^T (x-y)) dw
\\ &= \mathbb{E}_ {w}[\exp(i w^T (x-y))]
\\ &\approx \frac{1}{R} \sum_ {r=1}^{R} \exp(i w_r^T (x-y)) \\ &=
\begin{bmatrix}
\frac{1}{\sqrt{R}} \exp(i w_{1}^T x) \\ \frac{1}{\sqrt{R}} \exp(i w_{2}^T x) \\ \vdots \\ \frac{1}{\sqrt{R}} \exp(i w_{R}^T x)
\end{bmatrix}^T
\begin{bmatrix}
\frac{1}{\sqrt{R}} \exp(-i w_{1}^T y) \\ \frac{1}{\sqrt{R}} \exp(-i w_{2}^T y) \\ \vdots \\ \frac{1}{\sqrt{R}} \exp(-i w_{R}^T y)
\end{bmatrix}
\\ &= \mathbf{h}(x) \mathbf{h}(y)^{*} \end{align}
$$
为了避免虚数,我们定义:
$$
\begin{align}
w&\sim p(w)
\\
b&\sim Uniform(0,2\pi)
\\
z_w(x)&=\sqrt{2}cos(w^Tx+b)
\end{align}
$$
则
$$
\begin{align}
E_w(z_w(x)z_w(y))&=E_w[\sqrt{2}cos(w^Tx+b)\sqrt{2}cos(w^Ty+b)]
\\&=
E_w[cos(w^Tx+b)+2b]+E_w[cos(w^T(x-y))]
\\&=
0+E_w[cos(w^T(x-y))]
\end{align}
$$
故可定义:
$$
z(x) =
\left[\begin{matrix} \frac{1}{\sqrt{R}} z_{w_1}(x) \\ \frac{1}{\sqrt{R}} z_{w_2}(x) \\ \vdots \\ \frac{1}{\sqrt{R}} z_{w_R}(x) \end{matrix}\right]
$$
因此
$$
\begin{align}
z(x)^Tz(y)&=\frac{1}{R}\sum_{r=1}^R z_{w_r}(x)z_{w_r}(y)
\\
&=\frac{1}{R}\sum_{r=1}^R 2cos(w_r^Tx+b_r)cos(w_r^Ty+b_r)
\\
&=\frac{1}{R}\sum_{r=1}^R cos(w_r^T(x-y))
\\
&\approx E_w[cos(w_r^T(x-y))]
\\
&=k(x,y)
\end{align}
$$
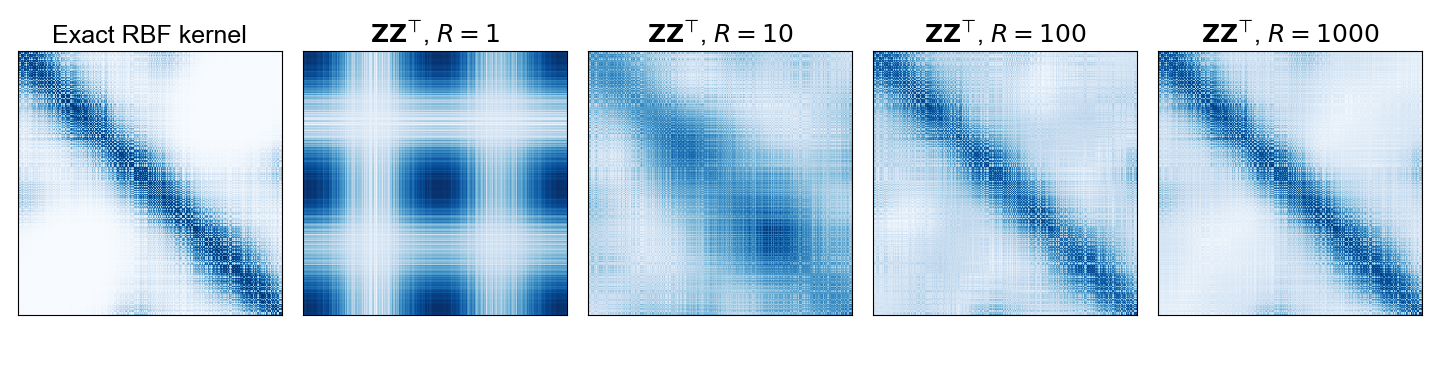
应用——高斯核近似
1 | |